- 服務
- ·
Nanopore circRNA全長測序
- 產品介紹
- 案例解析
- 結果展示
- 送樣建議
服務介紹
利用隨機引物對富集后的circRNA進行滾環反轉錄擴增,采用納米孔測序技術對circRNA的全長序列進行直接測序,并使用特定算法,實現對長測序片段中的circRNA序列進行識別和全長重構。
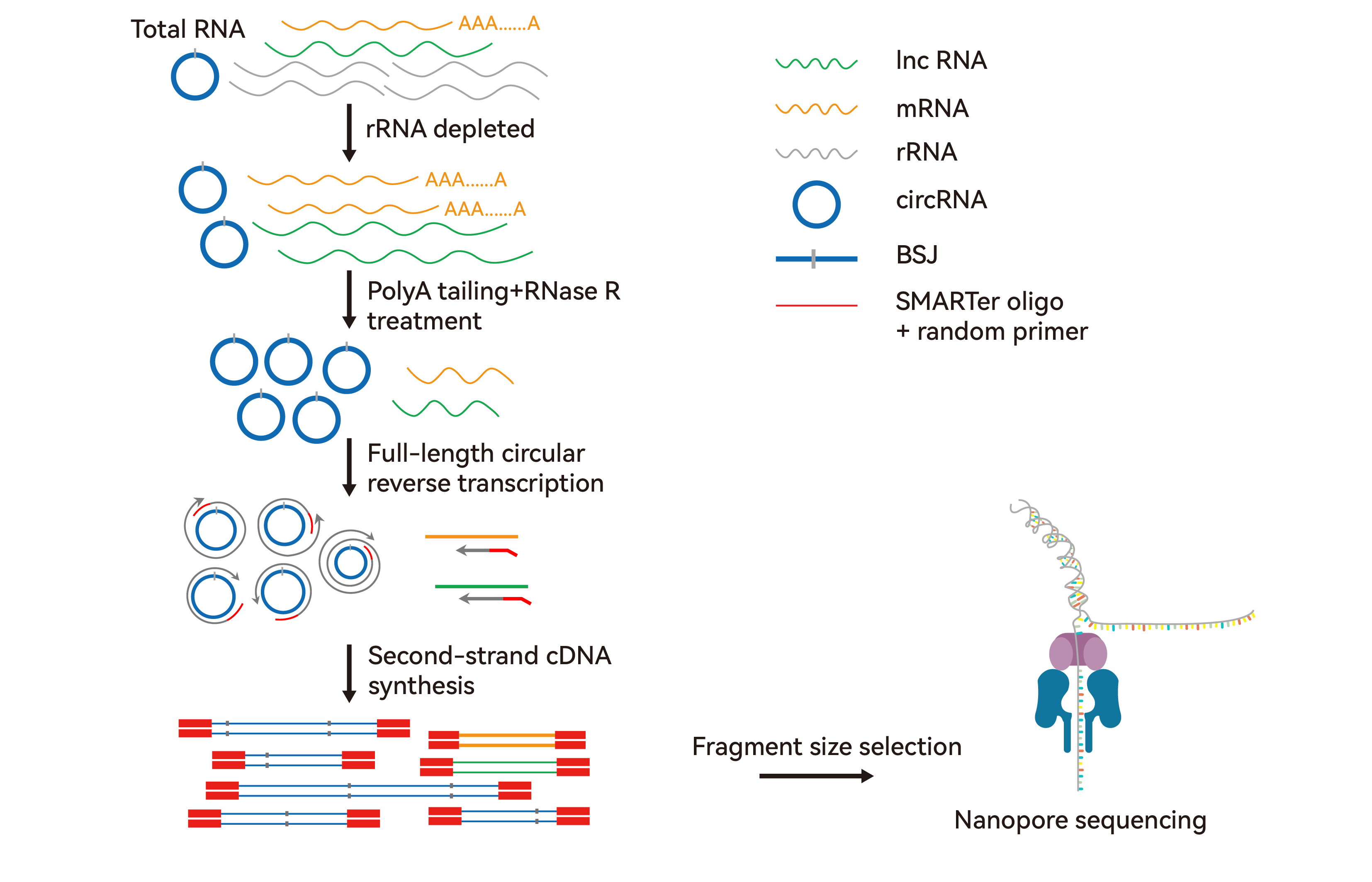
圖1:circRNA全長納米孔測序技術路線
測序方案
測序模式:Nanopore sequencing
測序數據量:1 Gb raw data
納米孔測序技術在檢測circRNA上的優勢:
1.更強的檢出率:相比二代測序,納米孔測序可以?將circRNA reads檢測效率提高20倍以上。
2.更高的靈敏度:可識別低豐度的circRNA,能夠更敏感地捕獲到非經典circRNA,i.e., intronic。
3.更多更準確的應用領域:
1)準確識別可變剪切事件,無需拼接,一鍵生成;
2)直接識別癌癥等重大疾病發生發展的重要生物標志物一一融合環狀RNA(f-circRNA);
3)能夠更敏感的識別研究熱點一一線粒體環狀RNA,確保您的研究不會落空。
基于三代測序技術分析circRNA全長
Comprehensive profiling of circular RNAs with nanopore sequencing and CIRI-long. Zhang J et al. Nature biotechnology, 2021. (IF: 36.558)
測序策略:circRNA Nanopore sequencing
研究樣本:小鼠腦組織
本研究開發了一種高效測定circRNA全長轉錄本的實驗和計算方法:利用隨機引物對circRNA進行的滾環反轉錄擴增后,使用納米孔測序技術對circRNA的全長序列進行直接測序,并開發了CIRI-long 算法,實現對長測序讀段中的circRNA序列進行準確識別和全長重構。
作者發現優化后的納米孔測序體系能夠在總Reads中測到6%的circRNA信息,而在傳統的PE150二代測序數據中,總RNA測序結果中只有0.06%的Reads是circRNA數據,RNase R處理也只能得到0.27% 的circRNA數據。相對于二代測序,納米孔三代測序結合CIRI-long分析方法可以將檢測效率提高20多倍。并且,納米孔三代測序能夠實現<100bp~5kb長度circRNA的全序列組裝,而二代測序(PE150)僅能拼接300bp以內的序列。比較對照的二代測序數據以及CircAtlas數據庫信息,本文的體系得到了更多circRNA信息。納米孔測序得到了一半多的全新circRNA,但這些circRNA的豐度較低,說明納米孔測序體系具有更高的靈敏度,可以檢測到更多的circRNA分子(圖1)。兩次生物學重復的樣品能很好地吻合,說明該體系具有較高的穩定性(圖2)。
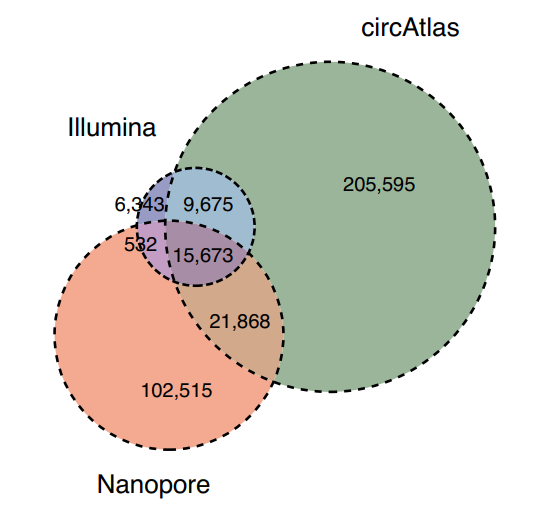
Figure 1. 納米孔測序體系具有更強的檢出率
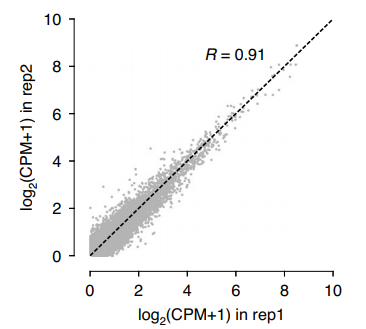
Figure 2. 納米孔測序體系具有較高的穩定性
circRNA可變剪切是多樣性的重要體現,作者從這批納米孔測序數據中分析了circRNA可變剪切的情況,在15905個基因來源的circRNA中共分析到115755種可變剪切分子。而二代測序的數據中只從6928個基因中分析到了25159種可變剪切分子。四種類型的可變剪切都存在,總體而言,在納米孔測序的數據中分析得到的可變剪切數目均高于二代測序的數據(圖3)。作者還發現了內含子自動形成的circRNA(圖4)。
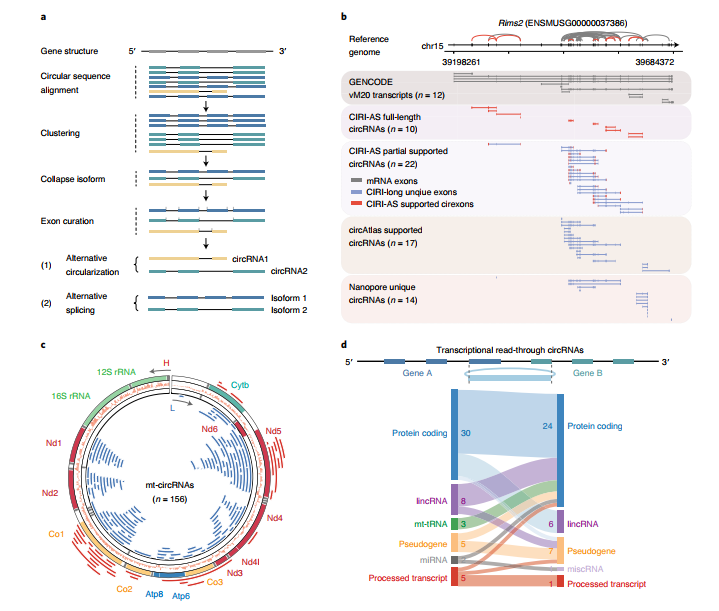
Figure 3. CIRI-long分析circRNA和剪切多樣性
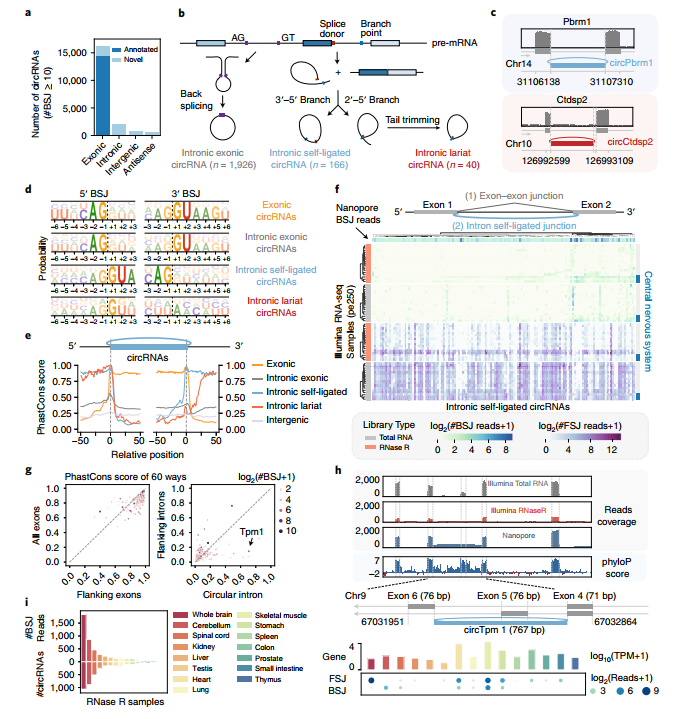
Figure 4.內含子自剪切形成的circRNA
生物信息分析
基礎分析
原始數據質控檢查
比對結果質控檢查
樣品表達質控
基于circRNA表達的樣品主成分分析
circRNA全長鑒定
circRNA在染色體上的分布特征分析
circRNA長度統計與分析
circRNA可變剪切isoform識別
高級分析
circRNA差異分析
差異circRNA宿主基因的GO功能分析
差異circRNA宿主基因的KEGG通路富集分析
差異circRNA宿主基因的Reactome通路富集分析
差異circRNA的靶向miRNA預測分析
circRNA及靶向miRNA網絡調控圖
circRNA-RBP 關系預測
circRNA 翻譯潛能預測
個性化分析
融合基因來源的f-circRNA識別融合基因來源的f-circRNA
部分結果示例
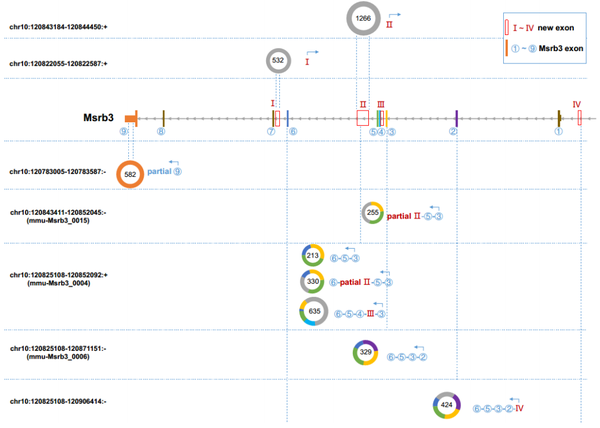
圖1:circRNA全長納米孔測序鑒定隱藏且復雜的轉錄與可變剪切圖譜
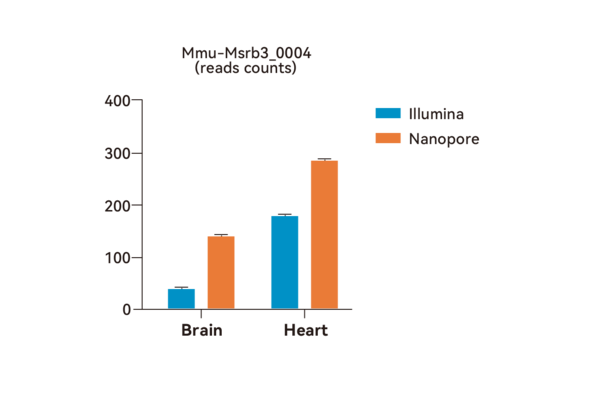
圖2:mmu-circMsrb3 0004在小鼠心臟/腦中的表達水平
小鼠基因Msrb3只包含3個線性轉錄本,卻能夠轉錄至少10個環狀轉錄本分子(如圖1)。circRNA全長納米孔測序鑒定出的circRNAs多種多樣,包括exonic circRNAs、ElciRNAs、intronic circRNAs,相比lllumina僅僅檢測BSJ,Nanopore全長鑒定能夠觀察到circRNA真實的內部構造,甚至發現新的circRNA外顯子(如圖1中紅色框)。
特別地,Nanopore以及lllumina都檢測到mmu-circMsrb3_0004在小鼠心臟/腦中有較高的表達(如圖2),circRNA全長納米孔測序更是識別到該環狀分子的3個isoforms。
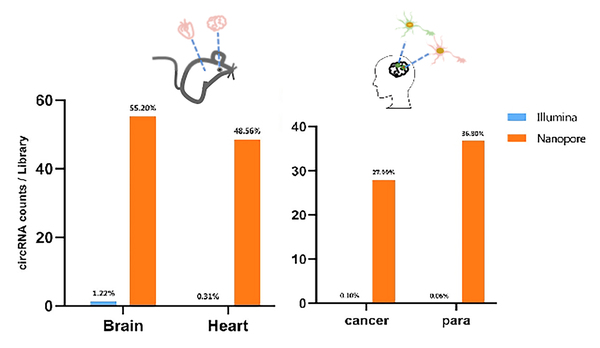
圖3:Nanopore與lllumina測序circRNA豐度比較
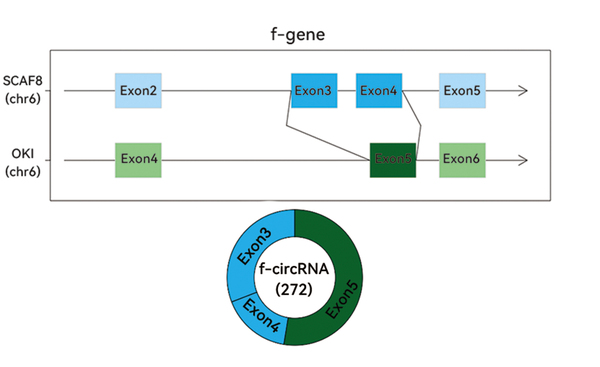
圖4:circRNA全長納米孔測序檢測到6號染色體的基因SCAF8的外顯子3-4與QKI的外顯子5發生了融合,并形成了環狀RNA分子。
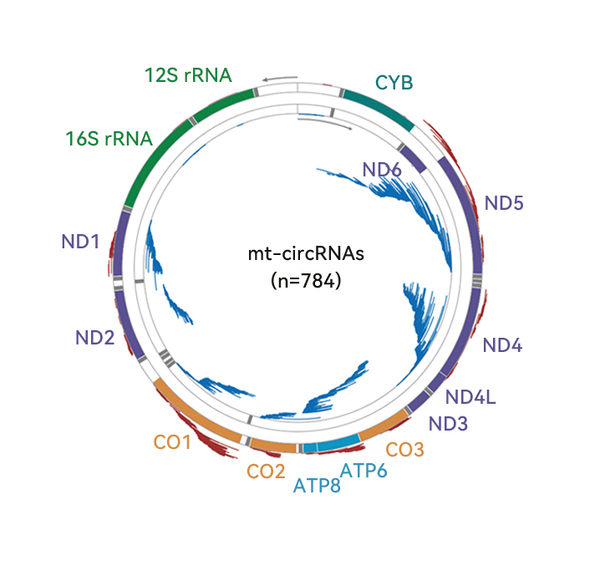
圖5:circRNA全長納米孔測序在人類前列腺癌細胞中檢測到了大量線粒體環狀RNA,這些分子主要分布于輕鏈。
Table.ONT circRNA full-length Seq原始樣本送樣建議
送樣類型
送樣量
備注
細胞 (細胞數)
≥2*10^6
無支原體污染
動物組織
≥200mg
植物組織
≥200mg
全血
≥4mL
Table.ONT circRNA full-length Seq RNA樣本送樣建議
送樣類型
送樣量
完整性(RIN值)
濃度
純度
total RNA(組織、細胞、全血等)
≥4μg
≥7
≥100ng/μl
無DNA,蛋白/鹽離子等污染,樣本無色透明不粘稠
*更具體的送樣方法請詳詢銷售或技術支持
物種范圍:人、小鼠、大鼠等哺乳動物,擬南芥、水稻、番茄、玉米、大豆等植物,其他物種詳詢銷售或技術支持
填寫需求描述給我們
工具快速咨詢
400-8989-400
geneseed@geneseed.com.cn

 購物車
購物車




 廣州市黃埔區開源大道11號科技企業加速器A區6棟2樓
廣州市黃埔區開源大道11號科技企業加速器A區6棟2樓


 geneseed@geneseed.com.cn
geneseed@geneseed.com.cn