- 產品
- ·
RNA pull-down試劑盒
- 產品介紹
- 案例分析
- 客戶文章
- 說明書下載
- Q&A

RNA pull-down是檢測RNA結合蛋白與RNA之間相互作用的主要實驗手段之一。該技術利用體外合成生物素標記RNA模擬天然RNA分子,作為探針,與細胞裂解液孵育。探針與裂解液中的蛋白形成RNA-蛋白質復合物,可被鏈霉親和素磁珠特異性結合捕獲,從而實現RNA結合蛋白(RBPs)的捕獲富集。
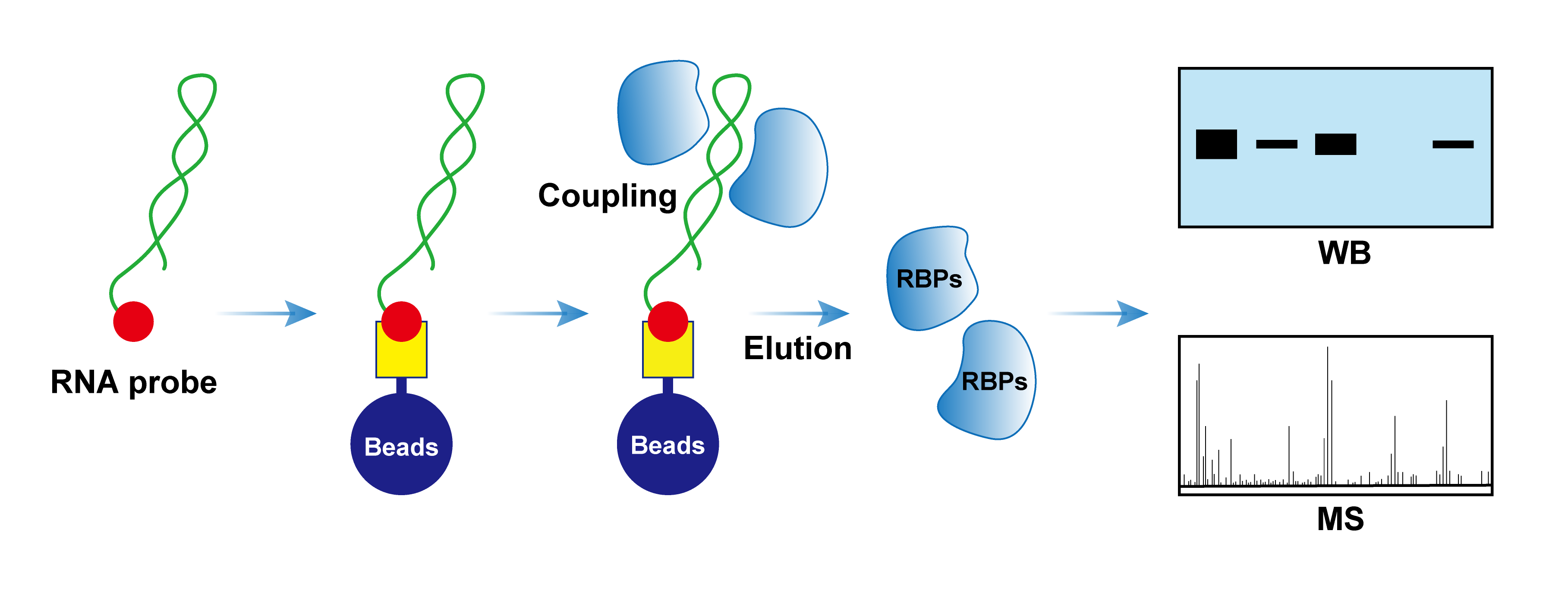
產品特點
1. 方便:試劑盒成分完整,包含pull-down實驗整套試劑;
2. 快捷:2.5小時即可完成;
3. 穩定:捕獲效率高,實驗體系穩定,重復性高。
產品應用
● 靶RNA結合蛋白的驗證或篩選;
● 適用長鏈、線性 RNA 探針(模擬靶 RNA 序列)直接拉取互作蛋白。
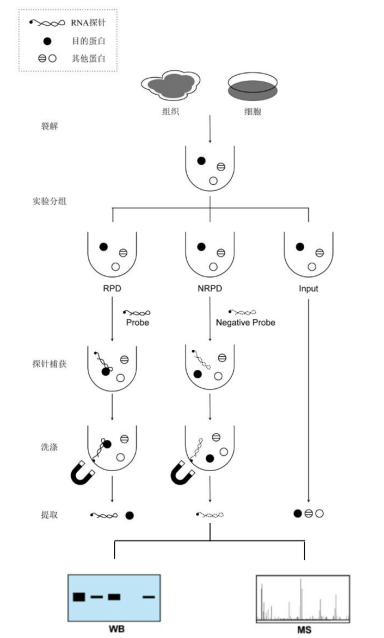
操作流程
結果展示
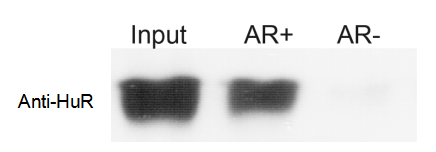
圖1. AR+探針特異性捕獲HuR蛋白
RNA pulldown 實驗就是先將RNA進行生物素標記,再與細胞裂解液共同孵育,從而形成RNA-蛋白質復合物,之后再分離復合物得到蛋白質,通過WB或質譜檢測蛋白質的技術。
我們知道,RIP是從蛋白出發研究蛋白RNA相互作用的技術,通過已知蛋白去驗證可能有相互作用RNA分子;而RNA pull-down則是從RNA出發研究蛋白與RNA相互作用的技術,通過已知RNA去拉蛋白,推測可能與已知RNA結合的蛋白分子。所以RIP和RNA pull-down兩種技術可以從正反雙向進行驗證,能更準確的反映RNA與蛋白的相互作用。
案例1

胃癌是全球第四大診斷類癌癥,也是全球癌癥相關死亡的第三大常見原因。由于胃癌進展的病理機制尚不完全清楚,因此需要更多的研究來發現和開發胃癌診斷和治療的有效生物標志物和靶標。在本文中,作者發現并命名了一種新的lncRNA_GClnc1。它作為支架(Scaffold) lncRNA募集WDR5和KAT2A這兩種蛋白質,并激活靶基因SOD2的轉錄,最終促進胃癌進展。
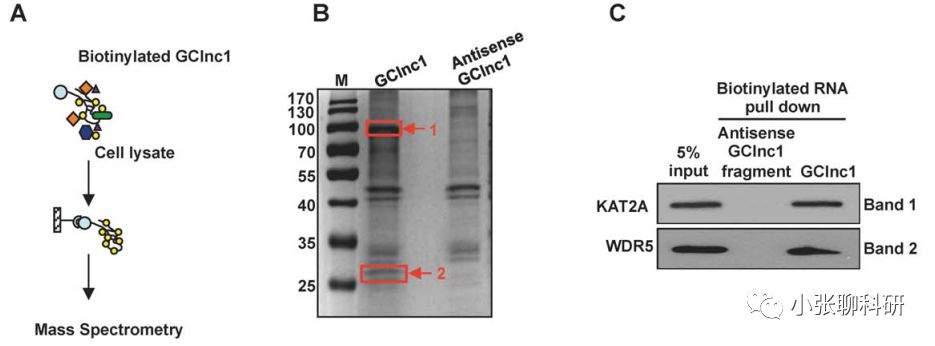
圖1. GClnc1 RNA pull-down結果圖
本文研究中,為了探索GClnc1促進胃癌的機制,作者使用了RNA pull-down分析來鑒定細胞內GClnc1結合因子(Fig-1A)。 GClnc1下拉樣本中有兩個特定的條帶。作者鑒定了94和101種潛在的結合蛋白(Fig-1B)。Western印跡顯示僅WDR5和KAT2A特異性結合GClnc1(Fig-1C)。數據表明這兩種蛋白質可能形成復合物以與GClnc1相互作用。[1]
案例2

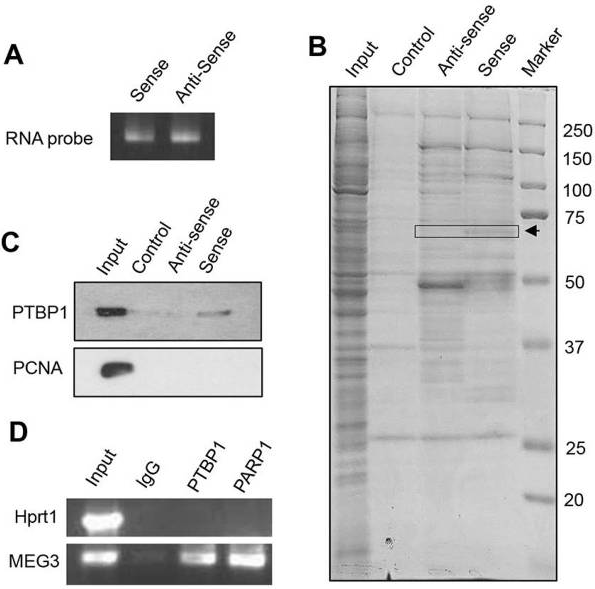
圖1 Pulldown & RIP揭示PTBP1蛋白與MEG3 RNA的相互作用
(A) 通過體外轉錄合成生物素標記的有義和反義 (陰性對照) MEG3 RNA。(B)體外RNA 下拉試驗。HEK293T 細胞裂解物與生物素標記的寡核苷酸一起孵育。下拉后,對蛋白質進行 SDS-PAGE 并用考馬斯亮藍染色。對箭頭指示的條帶進行質譜分析。(C) 蛋白質印跡以確定有義MEG3 RNA 與 PTBP1 蛋白的特異性相互作用,但不與 PCNA 蛋白(陰性對照)。(D) PTBP1 與MEG3 RNA在體內相互作用的 RNA 免疫沉淀 (RIP)在 HEK293T 細胞中。使用 MEG3 或 Hprt1 的特異性引物(陰性對照)通過 RT-PCR 測定由抗 PTBP1 或 IgG 免疫沉淀的蛋白質-RNA 復合物。
該研究中,首先通過RNA Pulldown和后續質譜實驗找到了與lncRNA MEG3結合的蛋白PTBP1,然后再通過PTBP1的抗體的RIP實驗反向驗證了lncRNA MEG3與PTBP1蛋白的結合。
案例解析參考文獻:
[1] Tian-Tian Sun, Jie He, Qian Liang, Lin-Lin Ren, Ting-Ting Yan, Ta-Chung Yu, Jia-Yin Tang, Yu-Jie Bao, Ye Hu, Yanwei Lin, Danfeng Sun, Ying-Xuan Chen, Jie Hong, Haoyan Chen, Weiping Zou and Jing-Yuan Fang. LncRNA GClnc1 Promotes Gastric Carcinogenesis and May Act as a Modular Scaffold of WDR5 and KAT2A Complexes to Specify the Histone Modification Pattern. Cancer Dis. July 2016
[2] Long noncoding RNA MEG3 induces cholestatic liver injury by interaction withPTBP1 to facilitate shp mRNA decay.Hepatology 2017 02;65(2)
[1] IF8.469 Linc-ROR facilitates progression and angiogenesis of hepatocellular carcinoma by modulating DEPDC1 expression. Cell Death & Disease
[2] IF6.684 Inhibition of miR-185-3p Confers Erlotinib Resistance Through Upregulation of PFKL/MET in Lung Cancers. Frontiers in Cell and Developmental Biology
[3] IF6.244 circDNMT1 Promotes Malignant Progression of Gastric Cancer Through Targeting miR-576-3p/Hypoxia Inducible Factor-1 Alpha Axis. Frontiers in Oncology
[4] IF5.555 Long Noncoding RNA X-Inactive-Specific Transcript Promotes the Secretion of Inflammatory Cytokines in LPS Stimulated Astrocyte Cell Via Sponging miR-29c-3p and Regulating Nuclear Factor of Activated T cell 5 Expression. Frontiers in Endocrinology
[5] IF5.502 Silencing of LINC01963 enhances the chemosensitivity of prostate cancer cells to docetaxel by targeting the miR-216b-5p/TrkB axis. LABORATORY INVESTIGATION
[6] IF5.458 Increased LDL receptor by SREBP2 or SREBP2-induced lncRNA LDLR-AS promotes triglyceride accumulation in fish. iScience
[7] IF5.241 The lncRNA MIAT regulates CPT-1a mediated cardiac hypertrophy through m6A RNA methylation reading protein Ythdf2. Cell Death Discovery
[8] IF5.228 C/EBPα promotes porcine pre-adipocyte proliferation and differentiation via mediating MSTRG.12568.2/FOXO3 trans-activation for STYX. BIOCHIMICA ET BIOPHYSICA ACTA-MOLECULAR AND CELL BIOLOGY OF LIPIDS
[9] IF4.599 CircDTL Functions as an Oncogene and Regulates Both Apoptosis and Ferroptosis in Non-small Cell Lung Cancer Cells. Frontiers in Genetics
[10] IF4.36 Circ_0001667 knockdown blocks cancer progression and attenuates adriamycin resistance by depleting NCOA3 via releasing miR-4458 in breast cancer. DRUG DEVELOPMENT RESEARCH
[11] IF3.575 Knockdown of circBFAR inhibits proliferation and glycolysis in gastric cancer by sponging miR-513a-3p/hexokinase 2 axis. BIOCHEMICAL AND BIOPHYSICAL RESEARCH COMMUNICATIONS
[12] IF2.952 Circular RNA OMA1 regulates the progression of breast cancer via modulation of the miR?1276/SIRT4 axis. Molecular Medicine Reports
[13] IF2.626 Regulation of circADAMTS6-miR-324-5p-PIK3R3 ceRNA pathway may be a novel mechanism of IL-1β-induced osteoarthritic chondrocytes. JOURNAL OF BONE AND MINERAL METABOLISM
[14] IF2.57 Long Non-Coding RNA GABPB1-AS1 Augments Malignancy of Glioma Cells by Sequestering MicroRNA-330 and Reinforcing the ZNF367/Cell Cycle Signaling Pathway. Neuropsychiatric Disease and Treatment
[15] IF2.192 Silencing of Circ_0135889 Restrains Proliferation and Tumorigenicity of Human Neuroblastoma Cells. JOURNAL OF SURGICAL RESEARCH
1、 這個pulldown試劑盒能做circRNA嗎?
目前公司的RNA-Protein pulldown試劑盒適用于線性RNA,circRNA可以了解標簽法,公眾號解讀“新一代circRNA pulldown 研究方法”有詳細的介紹。
2、 pulldown產物是否能檢測互作的RNA?用Trizol是否能抽提到RNA?
試劑盒體系中目前產物的分離體系主要是蛋白質,不涉及RNA提取及檢測。
3、 目的探針是正義鏈探針還是反義鏈?
目的探針是正義鏈探針,陰性對照是其反義鏈。
4、 探針的建議用量是多少?
探針的使用量推薦50pmol/次,或根據實驗具體情況調整用量。
5、 關于生物素標記探針
該試劑盒應用的生物素標記RNA探針,目的是模擬天然的RNA分子,理論上覆蓋的序列越全越好,可通過直接合成或體外轉錄方式獲取。但序列過長(大于2kb)則不易獲得,可優先選擇有預期蛋白結合的區域。
6、 怎么判斷實驗操作有沒有問題?
建議在實驗過程中設置陽性對照,試劑盒中是包含陽性對照探針和HuR抗體的;HuR蛋白幾乎存在所有細胞樣本中,且能夠被陽性探針拉下,結合力較強。可以通過陽性對照HuR的WB結果判斷實驗操作是否有問題,陰性對照可選擇性進行。
7、 pull down試劑盒中的loading buffer顏色很淡,和平時用的不一樣,是否正常?
我司pulldown試劑盒的loading buffer 確實偏淡一些。主要考慮loading buffer過多會對核酸的跑膠結果有一定影響,因此能看到條帶顯示就足夠了。
8、 RAP(RNA 反義純化)與RNA-protein pulldown有哪些異同?
相同點:都是探針拉取RNA結合復合物。
不同點:
1)RAP本質是反義探針拉取靶基因及其結合其上的RNA、蛋白甚至DNA等復合物。而RNA-protein pulldown是靶基因正義序列探針釣取結合蛋白。
2)2)RAP本質是反義探針與靶基因RNA的核酸雜交,類似FISH,反應溫度基本在37℃。而RNA-protein pulldown本質是長鏈核酸探針與蛋白之間的結合互作,反應溫度在4℃。
填寫需求描述給我們
工具快速咨詢
400-8989-400
geneseed@geneseed.com.cn
- 返回頂部

 購物車
購物車




 廣州市黃埔區開源大道11號科技企業加速器A區6棟2樓
廣州市黃埔區開源大道11號科技企業加速器A區6棟2樓


 geneseed@geneseed.com.cn
geneseed@geneseed.com.cn